R
|
| ||
| Area: | Estadística | |
| Web del proyecto: | r-project | |
Este artículo es demasiado largo. Si alguien se anima, debería segmentarse (ejemplo) y además usar la integración entre R y MediaWiki.
R es un software de análisis estadístico de código abierto basado en el lenguaje S, desarrollado a principio de los años 90. El programa $-Plus utiliza el mismo lenguaje S, pero se trata de un software propietario y con un precio prohibitivo para la mayoría de los mortales.
Si quieres, puedes consultar también nuestra guía sobre como poner en funcionamiento un completo sistema de trabajo para la investigación en medicina.
En la primera parte de este artículo (puntos 1 a 8) se muestran los principios generales de este software y la forma de instalar todos sus componentes. En la segunda parte (puntos 9 a 16) se explican algunas de sus principales funciones, con pequeños ejemplos para clarificar lo que se explica. Los puntos 17 y 18 se dedican a la exportación de resultados a distintos formatos. El punto 19 muestra un ejemplo tomado de la realidad y que ilustra algunos de los procedimientos expuestos. Finalmente en los puntos 20 a 22 se muestra como obtener ayuda adicional.
Instalación y ejecución
Sistemas M$
Los binarios para la instalación en sistemas M$ se pueden descargar desde aquí. Para entrar al programa hay que ejecutar el acceso directo a Rgui.exe situado en el menú inicio.
Debido al sistema de librerías que usa R, los cambios de versión pueden parecer complicados. Lo más sencillo es desinstalar la versión antigua, instalar la nueva y volver a bajar todas las librerías que se tuvieran instaladas. Sin embargo, también es posible conservar las carpetas correspondientes a las librerías que se quieran guardar, que se encuentran en la ruta:
c:/.../rw.../library
Hay que copiar dichas carpetas y pegarlas en el directorio donde instalemos la nueva versión.
Sistemas GNU/Linux
En Ubuntu la instalación se realiza mediante la aplicación Synaptic, buscando la librería r-base y aceptando la instalación de todas las librerías propuestas por el programa para resolver las dependencias. El programa se ejecuta desde una ventana Terminal tecleando simplemente
>R
Nota: ">" es el simbolo del sistema. asi que no hay que teclearlo
Si no se dispone de Synaptic o se quiere tener siempre la última versión del programa sin esperar a que aparezca en los repositorios, hay que bajar la versión correspondiente a nuestra distribución desde el CRAN.
La actualización a nuevas versiones puede realizarse igualmente desde Synaptic o desde el CRAN sin mayores problemas.
Lenguaje encarado a objetos
Los lenguajes estadísticos clásicos (SAS, Stata, SPSS) parten de unos datos a los que se aplican distintos procedimientos para obtener unos resultados.
R no actua de la misma forma. En R existen los denominados objetos, sobre los que se aplican funciones para transformarlos en otros objetos, sobre quienes, a su vez, pueden aplicarse nuevas funciones para obtener nuevos objetos y asi sucesivamente.
Una función es cualquier código de R al que se le pasan uno o más argumentos para transformar uno o más objetos.
Por lo tanto en R no existen propiamente datos y resultados, sino distintos tipos de objetos y una colección de operadores y funciones para modificarlos.
Una de las cosas que más cuestan de entender al principio es que R trabaja en columnas y no en filas como hacen los sistemas tradicionales. Para R cada columna o variable de una base de datos es un objeto, y en cambio las filas o casos no.
Los nombres de los objetos pueden contener mayúsculas, minúsculos, números y puntos. La única restricción es que deben empezar por una letra.
Si a un objeto no se le asigna ningún nombre, simplemente será impreso en pantalla y se perderá. Es muy interesante, pues, guardar con un nombre todos los objetos creados (u obtenidos como resultado de la aplicación de funciones sobre otros objetos) para poder usarlos así en cálculos posteriores, lo cual constituye la principal ventaja del lenguaje S sobre otros lenguajes estadísticos
Convenciones básicas de la sintaxis
R es de hecho un lenguaje de programación llamado S, y como tal no trabaja a través de menús como los sistemas estadísticos clásicos. Ello hace que sea fundamental el conocimiento de las reglas básicas de su sintaxis para poder usar luego correctamente las distintas funciones:
- Las mayúsculas y minúsculas representan distintos caracteres.
- Los espacios no representan nada y R los ignora.
- Los missings se representan por las siglas NA si los datos son numéricos, o por un espacio blanco si se trata de caracteres.
- El valor null indica que un objeto no contiene datos.
- El signo # provoca que la linea no se evalue, y por lo tanto permite realizar comentarios.
- Los decimales deben indicarse con el signo . .
- Los parentesis ( ) sirven para especificar los argumentos de las funciones.
- Los diferentes argumentos de una función deben separarse con , .
- Cuando un argumento de una función es una fórmula, se especifica con vardep~verindep1+varindep2... .
- Los corchetes [ ] sirven para especificar una posición dentro de un objeto.
- Los claudators {} se utilizan para abrir y cerrar procesos condicionales (if, for...), utilizados especialmente en la construcción de nuevas funciones.
- Todas las funciones son anidables, de tal forma que un argumento de una función puede ser a su vez una función.
- El signo $ se usa para referirse a una parte concreta y diferenciada de un objeto. Las distintas partes de un objeto pueden conocerse con la función names(nombreobjeto).
- Para asignar un nombre a un objeto se utiliza la combinación <-. Esta instrucción es fundamental.
El sistema de librerías
Una librería o paquete es un conjunto de funciones sobre una temática común. El sistema R-base trae las librerías diseñadas por el núcleo central de desarrolladores (core). Sin embargo, el modelo descentralizado de producción de R conlleva que muchas funciones interesantes no se instalen con el paquete básico sino que estén contenidas en paquetes o librerías externas que hay que instalar por separado. Aunque son descarganles desde muchos sitios de internet, se recomienda su descarga desde el repositorio Comprehensive R Archive Network (CRAN) porque se garantiza que su funcionamiento ha sido revisado y aprobado por el núcleo de desarrolladores.
Existen en R cuatro tipos de librerías:
- las que se instalan con el sistema R-base y se cargan por defecto al inicio (no requieren pasos adicionales para ser utilizadas)
- las que se instalan con el sistema R-base y no se cargan por defecto al inicio (requieren ser cargadas para ser utilizadas):
- las que no se instalan con el sistema R-base y hay que bajar del CRAN (requieren instalación desde CRAN y luego carga)
- las que no se instalan con el sistema R-base y no están disponible en el CRAN (disponibles en ficheros .zip que se encuentran habitualmente en páginas personales de internet, desde donde hay que descargarlas, instalarlas y luego cargarlas)
Nota: La nomenclatura utilizada para los tipos de librerias es propia y no estándar.
Instalación de librerias adicionales en sistemas M$ (tipos 3 y 4)
Desde el programa (Rgui) existe un menú para instalar paquetes directamente desde el repositorio CRAN (tipo 3) sin tenerlos que descargar previamente de forma manual.
Desde el menú también se pueden instalar paquetes a partir de ficheros .zip bajados manualmente. Suele tratarse de paquetes tipo 4, aunque también se puede descargar librerías tipo 3 de forma manual desde el CRAN y luego instalarlos como ficheros .zip.
Para actualizar todos los paquetes instalados (excepto el paquete básico), hay que usar la función correspondiente del menú (update packages).
Instalación de librerias adicionales en sistemas GNU/Linux. (tipos 3 y 4)
Al igual que el sistema base, algunos paquetes adicionales (tipo 3) de R pueden instalarse desde Synaptic en Ubuntu buscando la palabra CRAN.
Los paquetes se guardarán automáticamente en la ubicación:
/usr/lib/R/site-library/nombrepaquete
y estarán disponibles immediatamente para ser cargados cuando se desee.
Sin embargo, no todos los paquetes adicionales disponibles en el CRAN (tipo 3) se pueden descargar desde Synaptic, y hay muchas distribuciones GNU/Linux distintas de Ubuntu. En estos casos, si conocemos el nombre de la librería hay que teclear desde R (ejecutado en un Root terminal):
install.packages("nombrepaquete")
Con este procedimiento los nuevos paquetes se ubicarán en:
/usr/local/lib/R/site-library
Otra forma de proceder con análogos resultados es buscar directamente por navegador en el CRAN la librería que nos interesa, decargarla en formato .zip y luego desde R (en un Root terminal) escribir:
install.packages("rutacompleta/nombrearchivo.zip",CRAN=NULL)
Los paquetes de tipo 4 descargados en formato .zip se instalan también con la instrucción anterior en R bajo un Root terminal:
install.packages("rutacompleta/nombrearchivo.zip",CRAN=NULL)
Una forma alternativa de instalar los paquetes .zip sin necesidad de introducir órdenes de R es descomprimir todo su contenido en:
/usr/lib/R/site-library/nombrepaquete
o en:
/usr/lib/R/library/nombrepaquete
o en:
/usr/local/lib/R/site-library/nombrepaquete
Para actualizar todos los paquetes instalados (excepto el paquete básico), hay que teclear en R en un Root terminal:
update.packages()
Cargado de librerías
Cuando se inicia R, sólo las funciones contenidas en determinadas librerías del núcleo básico están disponibles para ser utilizadas (tipo 1) . Incluso algunas librerías del núcleo básico no están cargadas al iniciar para ahorrar recursos (tipo 2). Para cargar una librería (tipos 2, 3 y 4), hay que utilizar la función library(). Por ejemplo para cargar la librería 'survival' (para el análisis de supervivencia), que se instala con el paquete básico pero no se carga al inicio, habría que escribir:
library(survival)
Si no la descargamos antes, una librería permanecerá cargada hasta que salgamos del programa, así que sólo hay que cargarla una vez por sesión.
Para descargarla:
detach(package:nombrelibreria)
Algunas librerías útiles
Las siguientes librerias *(tipo 2) y **(tipo 3) no se cargan directamente al iniciar R, y contienen funciones que pueden resultar interesantes:
- survival*: Funciones de análisis de supervivencia.
- car**: Recodificación de variables.
- xtable**: Permite exportar tablas a formato LaTeX.
El paquete Traba (tipo 4)
En el Trabachat se han desarrollado un conjunto de funciones agrupadas en el paquete Traba. El link anterior permite su descarga en formato .zip para poderlo instalar.
Se puede descargar también el código fuente para ver o modificar la sintaxis de las distintas funciones que contiene.
Editores de sintaxis
El programa R (a diferencia de SPSS, SAS o Stata) no cuenta con una ventana para escribir la sintaxis.
Cualquier editor de textos, como el Notepad de M$, sirve para almacenar los comandos, pero es recomendable trabajar con otros editores específicos que permitan, entre otros:
- Colorear la sintaxis para una fácil detección de errores
- Enviar las instrucciones directamente para ser evaluadas en R (evitando asi tener que copiar/pegar cada instrucción desde el editor)
Cualquiera que sea el programa elegido, se aconseja guardar los ficheros de sintaxis con la extensión .R.
WinEdt
WinEdt es un software propietario, disponible sólo para plataformas M$, que permite la edición de distintos tipos de lenguajes, entre ellos Stata y R. Para poder utilizar el editor debe disponerse de licencia, y descargarse la librería de R RWinEdt, disponible en el CRAN. Hay que iniciar WinEdt desde R tecleando:
library(RWinEdt)
WinEdt permite colorear la sintaxis de R, marcando las funciones correctas, y coloreando los paréntesis para evitar errores de apertura y cierre. Así mismo permite mandar una región de la sintaxis para su evaluación en R.
GNU/Emacs y XEmacs
Emacs es un anciano y robusto software para la edición de distintos lenguajes, con la posibilidad de ejecutar distintos programas en sus buffers lo cual permite evaluar directamente las expresiones dentro del propio programa. Por lo tanto Emacs es mucho más que un editor y coloreador de sintaxis, en algunos casos es poco menos que un estilo de vida.
El desarrollo de Emacs se ha dividido principalmente en dos proyectos: el programa original GNU Emacs (mas en consonacia con el movimiento software libre) y una ramificación del mismo XEmacs (mas en consonancia con el movimiento OpenSource).
Para la edición de lenguajes estadísticos en cualquiera de estos 2 programas debe usarse un modo de Emacs llamado Emacs Speaks Statistics (ESS).
Para sistemas M$ es aconsejable la instalación de XEmacs, cuyo instalador binario puede descargarse directamente desde la página web del proyecto, pues permite incorporar directamente cualquier modo, entre ellos Emacs Speaks Statistics (ESS).
En sistemas GNU/Linux se aconseja la instalación de GNU Emacs por su mayor facilidad de integración con R en estos sistemas. En Ubuntu y a través de Synaptic se puede instalar fácilmente tanto GNU Emacs como el modo Emacs Speaks Statistics (ESS), sin necesidad de mayores configuraciones.
Una vez instalados Emacs y Emacs Speaks Statistics (ESS), hay que abrir con Emacs un arhivo de código de R con la extensión .R. Automáticamente Emacs detecta que se trata de código de R y muestra unos nuevos botones relacionados con este sistema. Para iniciar la ejecución de R dentro de Emacs hay que pulsar el primer botón, identificado con una R azul. Para visualizar el buffer de R hay que darle al último botón, el que muestra una flecha azul y el texto ESS. De esta forma deberíamos tener en la parte superior de la pantalla el fichero (buffer) con la sintaxis de R (si no es así, debemos seleccionarlo a través del menú Buffers), y en la parte inferior el buffer donde se está ejecutando R. Podemos mandar partes del código a R pulsando el cuarto botón del panel de R.
GUI's
El hecho que R no tenga los menús para seleccionar funciones como tienen algunos otros sistemas de análisis estadístico puede hacer que resulte complejo su uso al principio. Para ello se han desarrollado varios GUI (Graphical User Interface) que ayudan al principio a irse familiarizandose con el lenguaje, aún a costa de perder flexibilidad. Aquí se puede encontrar un resumen de los principales GUI para R. Algunos de ellos son los siguientes:
- Rcomander: Se instala a través de la librería Rcmdr (tipo 3). Multiplataforma.
- SciViewsR: Disponible desde el 2 de abril de 2005 (es un desarrollo en estado alfa, solo disponible para windows de momento) esta interfaz grafica de R se asemeja a la que utiliza el programa SAS que divide la ventana en 3 secciones (una para edición de sintaxis, una para visualizar resultados y una tercera para navegar por los objetos y funciones).
SciViews-R es un conjunto de paquetes para R (principalmente el editor de texto Tinn-R, R2HTML, el GUI Rcmdr y algunos mas) que se integran en una interfaz grafica para R
Requiere los siguientes pasos previos:
- el programa R instalado y funcionando, el Rgui debe estar en modo sdi (Edit/Gui preferences/sdi, y salvar) hay que cerrar y volver a abrir el programa para que se apliquen los cambios.
- descargar e instalar sciViews-R_0.7-3Setup.exe , aceptando los iconos en el escritorio y en el menu de inicio.
- una vez instalado tenemos que entrar en la carpeta donde se ha instalado SciViews y modificar las propiedades del acceso directo a sciViews-R (boton derecho del mouse/propiedades/ en donde dice destino debe quedar reflejado el camino a Rgui.exe y las opciones que se describen abajo así:
"C:\Archivos de programa\R\rw2010pat\bin\Rgui.exe" --sdi RSciViews.RData
Ten presente que tu camino correcto puede diferir de lo que se ejemplifica aqui.
En caso de estar detras de un proxy tambien hay que añadir la opción:
http_Proxy=http://direccion.del.proxy:puerto/
Que permitira que se pueda conectar a la red para descargar paqueteria necesaria.
ya se puede proceder a lanzar la aplicación desde el icono que ha quedado en el menu de inicio o en el escritorio (es el mismo icono que R pero con tres atomos de colores). a medida que se utilize puede que necesite mas paquetes (por ejemplo para cada función que se ha implementado bajo Rcmdr (editar la base de datos, ciertos test estadisticos, etc...) siguiendo un sistema de ventanas se escogera el repositorio (CRAN, Bioconductur, etc...) el mirror (Spain, Francia, etc...) y procedera a a la descarga e instalación de los mismos.
Tipos de datos
Hay distintos tipos de datos en R, algunos de los cuales son:
- logical: sólo permite los valores T o F.
- numeric: sólo pueden contener números reales.
- character: permite introducir tanto número como cualquier caracter.
Para saber qué tipo de datos contiene un objeto:
mode(nombreobjeto)
Tipos de objetos
Los distintos tipos de datos pueden estar contenidos en los muchos tipos distintos de objetos que existen en R. Para conocer de qué tipo es un objeto:
class(nombreobjeto)
En R hay muchos tipos de objetos y con muchas propiedades distintas. Para una aproximación más detallada, consultar los distintos manuales relacionados al final de este artículo. A modo de introducción, algunos tipos de objetos son:
Vectores
Cadenas unidimensionales (es decir una sola columna o fila) de un tipo único de valores (númericos, caracteres, etc.).
Hay distintas funciones para crear vectores, la más simple de la cuales es c(), que simplemente crea un vector a partir de los valores que se introducen:
c(1,2,3,4,5)
La instrucción anterior genera un vector que contiene 5 números (1 a 5), pero lo único que hará es imprimir en pantalla esta secuencia. Para guardar el vector como un objeto debemos escribir:
prueba<-c(1,2,3,4,5)
Ahora tenemos un objeto llamado prueba que contiene el vector de 5 números.
Una forma alternativa de obtener el mismo vector prueba es mediante una secuencia (sequence) de valores:
prueba<-seq(1:5)
La función seq permite especificar el primer valor, el último y optativamente la longitud del vector o los incrementos secuenciales entre el primer número y el último (por defecto uno).
Los vectores permiten, como todos los objetos de R, la realización de operaciones y funciones. Si por ejemplo ejecutamos:
prueba2<-prueba*2
obtendremos un objeto llamado prueba2 que corresponderá a un vector con los valores: 2, 4, 6, 8, 10.
La expresión:
prueba3<-prueba*prueba2
generará el vector prueba3 con los valores 2, 8, 18, 32, 50.
Para generar vectores con números repetidos se usa la función rep, que repetirá el primer argumento tantas veces como se especifique en el segundo:
prueba4<-c(seq(1:5),rep(3,7))
que generará un vector con los valores 1, 2, 3, 4, 5, 3, 3, 3, 3, 3, 3, 3.
Para extraer un valor concreto de un vector, por ejemplo el que esté en tercera posición:
prueba3[3]
devolverá por pantalla el valor 18. Lógicamente este valor se podría haber asignado a un objeto.
Para saber el número de valores de un vector:
length(prueba3)
Matrices
Estructura bidimensional donde todos los datos deben ser del mismo tipo.
Para crear una matriz debemos usar las siguientes funciones:
- column bind
prueba4<-cbind(rep(72,5),c(2,7,9,3,5))
generará la siguiente matriz por la unión de las dos columnas (cbind) especificadas: rep(72,5) (repite 5 veces el valor 72) y c(2,7,9,3,5)
| 72 | 2 |
| 72 | 7 |
| 72 | 9 |
| 72 | 3 |
| 72 | 5 |
Si queremos unir filas en lugar de columnas:
- row bind
prueba5<-rbind(seq(1:5),c(2,7,9,3,5))
| 1 | 2 | 3 | 4 | 5 |
| 2 | 7 | 9 | 3 | 5 |
Para recuperar un valor concreto de la matriz anterior, debe especificarse, como es habitual, dicha posición entre corchetes. En primer lugar se marca la posición de la fila y en segunda lugar la posición de la columna.
prueba6<-prueba5[2,3]
Con la función anterior, asignamos el valor 9 al objeto prueba6.
La función:
prueba7<-prueba5[2,3:5]
asignará al objeto prueba7 un vector con los valores 9, 3, 5, correspondientes a las columnas 3, 4 y 5 de la segunda fila.
Para conocer las dimensiones de una matriz usaremos:
dim(prueba5)
que devolverá el valor 2 (filas) 5 (columas).
Para asignar nombre a las filas y a las columnas:
dimnames(prueba5)<-list(c("Fila1","Fila2"),c("Col1","Col2","Col3","Col4",Col5"))
Factores
Este tipo de objeto es fundamental para el análisis estadístico pues es la forma como se tratan las variables categóricas.
Para crear un factor:
factor1 <-factor(c(1,1,1,2,2,3,3,3,3,2,2,1,1,2,2,1,1),labels=c("Bajo","Medio","Alto"))
La función:
mode(factor1)
devolverá numeric porque los datos introducidos son numéricos, pero si usamos la función:
class(factor1)
devolverá factor indicando que el objeto es del tipo Factor.
Para saber los distintos niveles de un factor, que equivaldrían a sus etiquetas en otros sistemas:
levels(factor1)
Listas
Las listas son objetos genéricos que corresponden a colecciones de distintos objetos que pueden ser de tipos distintos.
Data Frames
Como una matriz pero pudiendo contener distintos tipos de datos. Es lo que clásicamente se ha llamado base de datos.
Si disponemos por ejemplo de dos vectores (que se pueden entender como variables) y queremos unirlos en un data frame:
base<-as.data.frame(cbind(vector1,vector2))
Otros tipos de objetos
Muchas funciones de R producen como resultado un objeto de un tipo particular y único de esa función, aunque en realidad se trata de listas que contienen como componentes los disitntos apartados de los resultados. PAra referirse a cada una de las partes de una lista se usa el caracter $.
Por ejemplo la función glm, que ajusta modelos de regresión, produce un objeto de tipo glm que contiene los distintos resultados.
Captura de datos externos
La forma más habitual de obtener datos para su análisis en R es su captura desde otros formatos, habitualmente M$ Excel, SPSS o M$ Access.
Cuando R captura una base de datos externos, en la mayoría de los casos detectará correctamente el tipo de datos que contiene, asignando como factores las variables categóricas y como vectores las variables cuantitativas.
Las mayoría de formatos externos se pueden capturar con funciones contenidas en la librería foreign (tipo 2), mientras que para la captura de bases de datos relacionales (como M$ Access) es necesaria la libreria RODBC (tipo 3).
Captura de $P$$
El siguiente ejemplo muestra cómo capturar una hoja de datos de $P$$:
library(foreign)
read.spss("C:/rutacompleta/nombrearchivo.sav")
Esta instrucción, sin embargo, sólo nos imprimirá los datos en pantalla. Para conseguir capturar la base de datos como un objecto de R:
library(foreign)
base1<-read.spss("C:/rutacompleta/nombrearchivo.sav")
Así tendremos un objeto llamado base1 del tipo data.frame. Para saber qué variables contiene:
names(base1)
Para referirse a una variable en concreto de la base de datos:
base1$nombrevar
Para evitar tener que indicar cada vez el nombre del objeto base de datos para referirse a una de sus variables, podemos adjuntarlo al area de trabajo (workspace) usando la instrucción:
attach(base1)
Y entonces ya nos podremos referir a sus variables sin usar el prefijo de la base de datos:
nombrevar
Ya nos mostrará directamente el contenido de la variable nombrevar.
Captura de M$ Excel
R no permite capturar directamente los archivos .xls de M$ Excel. Para importar un archivo de M$ Excel debemos primero guardarlo con la extensión .dbf desde el propio M$ Excel, y luego ejecutar las funciones:
library(foreign)
base2<-read.dbf("C:/rutacompleta/nombrearchivo.dbf")
attach(base2)
Captura de M$ Access
El siguiente código muestra como capturar una tabla o una consulta desde una base de datos relacional creada con M$ Access:
library(RODBC)
canal<-odbcConnectAccess("c:/rutacompleta/nombrearchivo.mdb")
base4<-sqlQuery(canal,"select * from Hoja1")
attach(base4)
donde Hoja1 es el nombre de la tabla o consulta que se desea importar.
Transformación de datos
Los sitemas clásicos como SPSS poseen dos formas de crear nuevas variables, que genéricamente podemos denominar:
- compute cuando el objetivo es calcular el valor de nuevas variables (habitualmente cuantitativas) a partir de los valores de una o más variables ya existentes
- recode cuando el objetivo es asignar el valor a una nueva variable (hanitualmente categórica) a partir de los valores presentes en otras variables
La función compute no tiene un equivalente en R pues el sistema de asignación de objetos la hace innecesaria. Por ejemplo si quisieramos calcular la masa corporal a partir de la talla y el peso:
peso<-seq(45.5,85,.5) talla<-rep(seq(155,174),4) imc<-peso/(talla/100)**2
Para realizar el cálculo, por ejemplo, sólo en los hombres, puede usarse la función ifelse:
peso<-seq(45.5,85,.5) talla<-rep(seq(155,174),4) sexo<-rep(seq(1,2),40) imc<-ifelse(sexo==1,peso/(talla/100)**2,NA)
Nota: El doble signo igual indica que se está evaluando una función lógica. El signo igual único indica que se está asignando un valor.
La función recode, en cambio, sí que tiene su correspondiente función en R. Se trata de la función recode del paquete car (tipo 3). Por ejemplo, para recodificar la variable cuantitativa edad en la variable categórica edadc:
library(car) edad<-seq(45.5,85,.5) edadc<-recode(edad,"lo:55=1;55:65=2;65:75=3;75:hi=4",as.factor.result=T)
Nota: La expresión se evalua de izquiera a derecha y caso por caso. Si un caso satisface por ejemplo la primera condición (lo:55), se le asigna un 1 a edadc y se pasa al siguiente caso. Por lo tanto un caso de edad=55 tendrá una edadc=1 y no una edadc=2.
La transformación anterior se habría podido realizar también con un ifelse:
edad<-seq(45.5,85,.5) edadc<-ifelse(edad<=55,1,ifelse(edad>55 & edad<=65,2,ifelse(edad>65 & edad<=75,3,4)))
Para etiquetar variables se usa la función label del paquete Hmisc (tipo3):
library(Hmisc) label(nombrevariable)<-"etiqueta"
Para etiquetar los distintos valores de las variables categóricas (factores):
nombrefactor<-factor(nombrefactor,labels=c("valornivel1","valornivel2"))
Manipulación de bases de datos
La función más simple para transformar una base de datos es la función subset, que creará un nuevo objeto que contendrá sólo parte de los datos del Data Frame original:
hombres<-subset(base1,sexo==1)
Sin embargo, hay muchas otras funciones más complejas para la manipulación de bases de datos. Destacaremos tres de ellas:
- aggregate: sirve parar conseguir una base de datos con calculos agregados, segun los segmentos definidos por un identificador (segmenta segun el identificador, calculos agregados, colapsa segun el identifcador). de tal manera que se tendra un registro por cada valor univoco del identificador. su utilización se demuestra en el ejemplo del final del artículo.
- merge: permite unir dos bases de datos con distintas variables y un identificador común.
- reshape: permite estirar o engordar una base de datos. Se usa habitualmente en estudios de seguimiento donde se tienen los datos en vertical (un registro por visita) y quieren tenerse en horizontal (un registro por individuo).
Se recomienda consultar la ayuda correspondiente a dichas funciones para comprender correctamente su funcionamiento y todos los argumentos que pueden aceptar. Dos de ellas, aggregate y merge, se utilizan en el ejemplo que se presenta al final de este artículo.
Exploración de datos
Variables cuantitativas
R tiene distintas funciones para describir variables cuantitativas:
media<-mean(nombrevariable) mediana<-median(nombrevariable) desviacion<-sd(nombrevariable) maximo<-max(nombrevariable) minimo<-min(nombrevariable)
y muchas otras.
Para replicar los resultados de cualquiera de las funciones anteriores para distintos valores de una segunda variable que habitualmente será categórica (factor):
medias<-tapply(nombrevariablecuantitativa,nombrefactor,mean)
La prueba de normalidad de Shapiro-Wil se obtiene mediante:
shapiro.test(nombrevariable)
Para conocer el número de missings de una determinada variable podemos crear un subset de la variable que contenga sólo los valores missings (NA), y luego contar cuantos valores contiene ese subset:
length(subset(var,is.na(nombrevariable)==T))
La función Explore del paquete Traba devuelve todos estos índices y algunos más, de forma parecida al procedimiento Explore de SPSS.
Variables categóricas
Una variable categórica, o un Factor en el lenguaje de R, se explora mediante una distribución de frecuencias.
Para obtener el conteo de las distintas categorías de una variable usaremos:
tabla1<-table(nombrevariable)
Para obtener los porcentajes, usaremos:
prop.table(tabla1)
o para crearlo directamente desde la variable original:
prop.table(table(nombrevariable))
Para crear tablas de contingencia se usa el mismo sistema. Con:
tabla2<-table(nombrevariable1,nombrevariable2)
obtenemos los conteos correspondientes a cada una de las celdas.
Para saber los porcentajes:
prop.table(tabla2)
o directamente:
prop.table(table(nombredevariable1,nombredevariable2))
Esta función nos dará el porcentaje de cada celda respecto al total. Para obtener el porcentaje respecto al total de la cada fila:
prop.table(table(nombredevariable1,nombredevariable2),1)
Y el porcentaje respecto al total de cada columna:
prop.table(table(nombredevariable1,nombredevariable2),2)
Si asignamos las funciones anteriores a objetos, podremos capturar un valor concreto que nos pueda interesar:
prop1<-prop.table(table(nombredevariable1,nombredevariable2),2) valor21<-prop1[2,1]
o directamente:
valor21<-prop.table(table(nombredevariable1,nombredevariable2),2)[2,1]
A continuación se detalla un ejemplo real de como obtener una tabla de contingencia a partir de dos factores:
var1<-factor(c(1,1,1,1,1,3,3,3,3,3,2,2,2,2,1,1,1,2,2,3,3,1),labels=c("Bajo","Medio","Alto"))
var2<-factor(c(1,2,2,2,1,1,1,2,2,2,1,1,1,1,1,2,2,2,1,1,2,1),labels=c("Hombre","Mujer"))
prop.table(table(var1,var2),2)
De esta forma obtendremos una tabla con el sexo en las columnas y el nivel socio-económico en las filas, y en cada celda habrá el porcentaje sobre el total de la columna.
Pruebas estadísticas clásicas
t de Student
La comparación de medias mediante la prueba de t de student se realiza con la función t.test, cuya sintaxis es la siguiente:
t.test(variablecuantitativa~variablebinaria)
Por ejemplo:
cuanti<-c(3,3,4,4,6,6,8,8,9,10,1,1,5,21,23,4,4,5,65,12) catego<-c(2,1,1,1,2,2,1,1,1,1,1,1,2,2,2,1,1,2,1,2) t.test(cuanti~catego)
Si en lugar de imprimir el resultado del test por pantalla, lo que queremos es guardarlo como un objeto:
test<-t.test(cuanti~catego)
Ejecutando:
names(test)
tendremos una relación de todas las partes del test. Si, por ejemplo, nos interesa solo el valor p:
p<-test$p.value
o directamente:
p<-t.test(cuanti~catego)$p.value
De esta forma, el objeto p contiene el valor p de la prueba, y lo podemos utilizar para realizar cálculos posteriores.
Ji-cuadrado
La prueba de Ji-cuadrado debe aplicarse sobre tablas de conteos creadas con los procedimientos explicados anteriormente. Por ejemplo, para obtener el valor del estadístico ji-cuadrado y guardarlo en el objeto ji:
nivel<-factor(c(1,1,1,1,1,3,3,3,3,3,2,2,2,2,1,1,1,2,2,3,3,1),labels=c("Bajo","Medio","Alto"))
sexo<-factor(c(1,2,2,2,1,1,1,2,2,2,1,1,1,1,1,2,2,2,1,1,2,1),labels=c("Hombre","Mujer"))
ji<-chisq.test(table(nivel,sexo))$statistic
Para redondear el resultado a dos decimales:
round(ji,2)
Medidas de asociación
Las funciones risc y riscagr del paquete Traba calculan las medidas de asociación (OR y RR) a partir de datos individuales o de tablas de contingencia introducidas directamente:
library(traba) risc(nivel,sexo) riscagr(7,5,12,3)
Otras pruebas
Puesto que habitualmente se conoce el nombre de la prueba que se desea aplicar, se recomienda buscar la función que debe emplearse y sus argumentos en el sistema de ayuda, tal como se indica en el apartado correspondiente.
Algunas de estas funciones son:
corr(variablescuantitativas) #Matriz de correlaciones, puediendo especificar si se quiere Person o Spearman wilcoxon.test kruskal.test
Y muchas otras.
Regresión
R posee una potente función llamada glm que permite ajustar casi cualquier modelo lineal generalizado. Los argumentos mínimos a especificar son la fórmula del modelo, la família a la que pertenece y la función de linkado:
glm(vardep~varindep1+varindep2+varindep3,family(link))
La siguiente tabla muestra las famílias y links más comúnmente utilizados en ciencias de la salud:
| Modelo | Familia | Linkado |
|---|---|---|
| Lineal | gaussian | identity |
| Logística | binomial | logit |
| Poisson | poisson | log |
| Logbinomial | binomial | log |
Para ua relación completa se puede consultar:
?family
El análisis de supervivencia, incluidos los modelos de regresión paramétricos y el modelo de riesgos proporcionales de Cox no se ajustan con la función glm sino con las funciones específicas contenidas en el paquete survival (tipo 2):
library(survival) survreg() coxph()
Dichas funciones se acompañan de adecuadas ayudas para su uso.
El modelo de regresión binomial negativa tampoco se ejecuta mediante la instrucción glm sino con una instrucción específica contenida en el paquete MASS (tipo 2):
library(MASS) glm.nb()
En cualquier modelo de regresión, las variables independientes cuantitativas se introducen como tales en el modelo. Las variables categóricas se introducirán como tales siempre que estén definidas como factors, cosa que ocurrirá en la mayoría de ocasiones.
Así, por ejemplo, podemos realizar una regresión logística:
incontinencia<-c(0,1,1,1,1,0,0,0,1,0,0,0,1,0,0,1,0)
edad<-seq(50,75,length=17)
nivelsocio <-factor(c(1,1,1,2,2,3,3,3,3,2,2,1,1,2,2,1,1),labels=c("Bajo","Medio","Alto"))
modelo<-glm(incontinencia~edad+nivelsocio,family=binomial(logit))
Las dummys para las variables factor se crean automáticamente en todos los modelos de regresión, y por defecto se usa la categoría inferior como basal. Para comprobarlo:
contrasts(nivelsocio)
Para usar, por ejemplo, la categoría más alta como referencia:
contrasts(nivelsocio)<-contr.treatment(3,3)
donde el primer tres indica que el factor tiene tres niveles, y el segunda tres indica que hay que usar la tercera categoría como referencia. Para comprobarlo:
contrasts(nivelsocio)
A partir de este momento, cuando introduciéramos el factor nivelsocio en un modelo de regresión, su usaría esta codificación.
Los resultados de un modelo se almacenan en un objeto de tipo glm, al que en este caso hemos llamado modelo. Si no le hubieramos asignado nombre, los resultados se hubieran impreso por pantalla pero no habriamos podido trabajar con ellos.
Ejecutando:
names(modelo)
Podemos ver toda la lista de resultados que contiene el objeto. Si nos interesan los coeficientes:
modelo$coefficients
O más específicamente, si queremos el coeficiente de la variable edad:
modelo$coefficients[2]
Para imprimir todos los resultados:
modelo
O para que quede más bonito:
summary(modelo)
La expresión:
names(summary(modelo))
nos muestra todos los resultados que podemos obtener a partir de la función summary de un objeto glm, que son distintos de los que se obtienen a partir del objeto en sí. En este caso, para obtener el coeficiente de la variable edad deberíamos usar:
summary(modelo)$coefficients[2,1]
Los objetos de tipo glm pueden usarse com argumentos de las funciones interval y llh, ambas contenidas en el paquete Traba y que permiten calcular, respectivamente, los intervalos de confianza de los coeficientes y la prueba de razón de verosimilitud:
library(traba) interval(modelo) llh(modelo)
Gráficos
R es probablemente el sistema más potentes y flexible de creación de gráficos que existe actualmente. Existen tantas opciones que es imposible resumirlas en un espacio reducido.
La principal función para la creación de gráficos es plot, por lo que se aconseja consultar su ayuda cuando se quiera crear un gráfico:
?plot
y para consultar los múltiples parámetros que se pueden pasar a un gráfico:
?par
La función plot se denomina de alto nivel porque cualquier gráfico debe empezar con esta instrucción y sus argumentos indispensables. Esto abrirá una ventana de gráfico y empezará su creación.
Existen además muchas funciones gráficas denominadas de bajo nivel que van a modificar el gráfico presente en una ventana gráfica generada previamente por la función plot.
Además R permite guardar directamente un gráfico generado en un archivo de imagen para su uso posterior. Esto se puede hacer, por ejemplo, con la función jpeg.
Uno de los argumentos de la instrucción genérica plot es el tipo de gráfico que se desea crear. Sin embargo, para facilitar la creación de gráficos (aún a cuesta de perder flexibilidad) existen muchas funciones que crean gráficos concretos, como por ejemplo barplot, hist o boxplot:
boxplot(nombrevariable,color="orange")
La instrucción anterior creará un boxplot de color naranja de la variable nombrevariable
A continuación se muestran algunos ejemplos reales de gráficos creados en R, sin comentar el código::
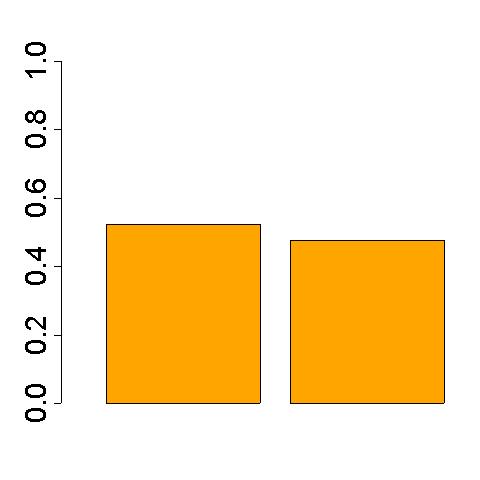
- Ejemplo 1: Gráfico de barras con el sexo de los pacientes alérgicos al huevo vacunados con triple vírica:
jpeg("c:/documents and settings/acrida/escritorio/TV Ou/gràfics/sexe.jpg")
barplot(prop.table(table(sexe)),ylim=c(0,1),col="orange",width=.3,xlim=c(0,.7),names.arg=F,cex.axis=1.8)
dev.off()
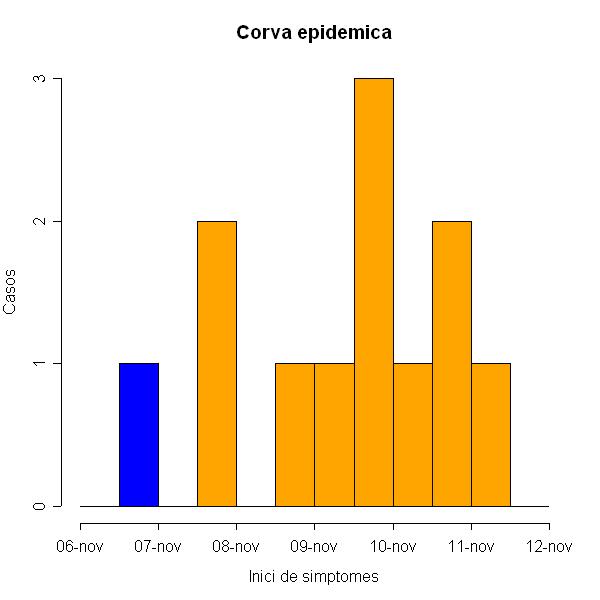
- Ejemplo 2: Curva epidémica (histograma) de una toxinfección alimentaria:
jpeg("t:/rs_cdp/Epidemiologia/francesc/Brots/GEA SS Hospi/corba.jpg",width = 600, height = 600)
hist(base,breaks=seq(ISOdatetime(2004,11,6,0,0,0),ISOdatetime(2004,11,12,0,0,0),by="12 hours"),right=F,freq=T,col="orange",axes=F,xlab="Inici de simptomes",ylab="Casos",main="Corba epidemica")
axis.POSIXct(base,side=1,at=seq(ISOdatetime(2004,11,6,0,0,0),ISOdatetime(2004,11,12,0,0,0),by="days"),format="%d-%b")
axis(side=2,at=seq(0,3,by=1))
polygon(c(ISOdatetime(2004,11,6,12,0,0),ISOdatetime(2004,11,6,12,0,0),ISOdatetime(2004,11,7,0,0,0),ISOdatetime(2004,11,7,0,0,0)),c(0,1,1,0),col="blue")
dev.off()
Programación de funciones
Otro punto fuerte de R es la posibilidad de crearse funciones propias, como es el caso de las contenidas en el paquete Traba. A continuación se explica el procedimiento de creación de una función mediante el ejemplo de las funciones interval y montehall de dicho paquete,
El código completo de ambas funciones puede encontrarse en el código fuente del paquete Traba.
El ejemplo de la función interval
Esta función va a calcular los intervalos de confianza para los parámetros de distintos modelos de regresión. Aunque existen funciones parecidas en otros paquetes, la intención al crear la función fue centralizar en una única expresión los cálculos para cualquiera de los modelos más usada en ciencas de la salud. Y aprender, claro :)
El primer paso consiste en crear un fichero con el nombre interval.R con cualquier editor (Emacs, WinEdt o cualquier editor de textos).
A continuación se irán mostrando las distintas partes del código con una breve descripción de los procedimientos que realiza, para que cada cual vaya descubriendo por si sólo la funcionalidad y sintaxis de las distinas partes:
1-Definimos el nombre de la función, los parámetros que deberá contener y sus valores por defecto en caso que los haya:
interval<-function(model,digits=2,nivell=0.95,alfa=F) {
2-Creamos algunas variables internas a partir de los parámetros recibidos, entre ellas el nivel de confianza a partir del alfa recibido
nivell2<--round(qnorm((1-nivell)/2),digits=2) co<-summary(model)$coef tot<-c()
3-Comprobamos que estamos recibiendo un modelo de regresión válido:
if(class(model)[1]!="lm" & class(model)[1]!="glm" & class(model)[1]!="coxph")
{
ex.warn <- options()$warn
options(warn = 0)
warning("El objeto no es un modelo de regresion valido",call.=F)
options(warn = ex.warn)
}
4-Cálculos si hemos recibido un modelo de regresión lineal:
if(class(model)[1]=="lm")
{
df<-model$df
p<-(1-nivell)/2
punt<-round(co[,1],digits=digits)
inf<-round(confint(model,level=nivell)[,1],digits=digits)
sup<-round(confint(model,level=nivell)[,2],digits=digits)
tot<-cbind(punt,inf,sup)
dimnames(tot)<-list(row.names(summary(model)$coef),c("B","inf","sup"))
}
5-Cáculos si hemos recibido un modelo de riesgos proporcionales de Cox:
if(class(model)[1]=="coxph")
{
punt<-round(exp(co[,1]),digits=digits)
inf<-round(exp(co[,1]-nivell2*co[,3]),digits=digits)
sup<-round(exp(co[,1]+nivell2*co[,3]),digits=digits)
tot<-cbind(punt,inf,sup)
dimnames(tot)<-list(row.names(summary(model)$coef),c("HR","inf","sup"))
alfa<-T
}
6-Cálculos si hemos recibido un glm (logística, logbinomial, poisson):
if(class(model)[1]=="glm")
{
if(summary(model)$family[1]=="gaussian")
{
ex.warn <- options()$warn
options(warn = 0)
warning("Utiliza la funcion lm en lugar de glm para los modelos de regresion lineal",call.=F)
options(warn = ex.warn)
}
else
{
punt<-round(exp(co[,1]),digits=digits)
inf<-round(exp(co[,1]-nivell2*co[,2]),digits=digits)
sup<-round(exp(co[,1]+nivell2*co[,2]),digits=digits)
tot<-cbind(punt,inf,sup)
if(summary(model)$family[1]=="binomial" & summary(model)$family[2]=="logit")
{
dimnames(tot)<-list(row.names(summary(model)$coef),c("OR","inf","sup"))
}
if(summary(model)$family[1]=="binomial" & summary(model)$family[2]=="log")
{
dimnames(tot)<-list(row.names(summary(model)$coef),c("RR","inf","sup"))
}
if(summary(model)$family[1]=="poisson")
{
dimnames(tot)<-list(row.names(summary(model)$coef),c("IRR","inf","sup"))
}
}
}
7-Preparamos e imprimimos los resultados:
if(!is.null(tot) & alfa==T)
{
return(tot)
}
if(!is.null(tot) & alfa==F)
{
tot<-tot[2:dim(tot)[1],]
if(length(tot)>3)
{
return(tot)
}
else
{
tot<-t(tot)
row.names(tot)<-row.names(summary(model)$coef)[2]
return(tot)
}
}
}
A continuación se presenta el código completo. Su edición con Emacs o WinEdt facilita su comprensión al colorear las funciones y los parentésis y claudators.
interval<-function(model,digits=2,nivell=0.95,alfa=F) {
nivell2<--round(qnorm((1-nivell)/2),digits=2)
co<-summary(model)$coef
tot<-c()
if(class(model)[1]!="lm" & class(model)[1]!="glm" & class(model)[1]!="coxph")
{
ex.warn <- options()$warn
options(warn = 0)
warning("El objeto no es un modelo de regresion valido",call.=F)
options(warn = ex.warn)
}
if(class(model)[1]=="lm")
{
df<-model$df
p<-(1-nivell)/2
punt<-round(co[,1],digits=digits)
inf<-round(confint(model,level=nivell)[,1],digits=digits)
sup<-round(confint(model,level=nivell)[,2],digits=digits)
tot<-cbind(punt,inf,sup)
dimnames(tot)<-list(row.names(summary(model)$coef),c("B","inf","sup"))
}
if(class(model)[1]=="coxph")
{
punt<-round(exp(co[,1]),digits=digits)
inf<-round(exp(co[,1]-nivell2*co[,3]),digits=digits)
sup<-round(exp(co[,1]+nivell2*co[,3]),digits=digits)
tot<-cbind(punt,inf,sup)
dimnames(tot)<-list(row.names(summary(model)$coef),c("HR","inf","sup"))
alfa<-T
}
if(class(model)[1]=="glm")
{
if(summary(model)$family[1]=="gaussian")
{
ex.warn <- options()$warn
options(warn = 0)
warning("Utiliza la funcion lm en lugar de glm para los modelos de regresion lineal",call.=F)
options(warn = ex.warn)
}
else
{
punt<-round(exp(co[,1]),digits=digits)
inf<-round(exp(co[,1]-nivell2*co[,2]),digits=digits)
sup<-round(exp(co[,1]+nivell2*co[,2]),digits=digits)
tot<-cbind(punt,inf,sup)
if(summary(model)$family[1]=="binomial" & summary(model)$family[2]=="logit")
{
dimnames(tot)<-list(row.names(summary(model)$coef),c("OR","inf","sup"))
}
if(summary(model)$family[1]=="binomial" & summary(model)$family[2]=="log")
{
dimnames(tot)<-list(row.names(summary(model)$coef),c("RR","inf","sup"))
}
if(summary(model)$family[1]=="poisson")
{
dimnames(tot)<-list(row.names(summary(model)$coef),c("IRR","inf","sup"))
}
}
}
if(!is.null(tot) & alfa==T)
{
return(tot)
}
if(!is.null(tot) & alfa==F)
{
tot<-tot[2:dim(tot)[1],]
if(length(tot)>3)
{
return(tot)
}
else
{
tot<-t(tot)
row.names(tot)<-row.names(summary(model)$coef)[2]
return(tot)
}
}
}
Una vez ejecutada la función, ya se puede utilizar:
interval(glm(vardep~varindep1+varindep2))
Para no tener que ejecutar cada vez la función, se recomienda incluirla en un paquete (como se hizo con el paquete Traba). El procedimiento para hacerlo se explica más abajo.
El ejemplo de la función montehall
Esta función reproduce la paradoja de Monte Hall. Se trata de un concursante que está en un concurso donde hay tres puertas. Detrás de una de ellas hay un coche y detrás de las otras dos una cabra (una en cada una de las otras dos puertas). Se supone que al concursante le hace más ilusión ganar el coche que la cabra. El presentador le hace escoger una puerta, pero antes de abrirla lo que hace es abrir una de las otras dos puertas (teniendo en cuenta que el presentador sabe dónde está el coche y abre siempre una puerta con una cabra). Luego le pregunta al concursante qué quiere hacer, si quedarse en la puerta que ha escogido o cambiar a la otra puerta que queda cerrada.
Intuitivamente la gente suele decir que es indiferente pues hay un 50% de probabilidad de acertar pues quedan dos puertas. Pero se demuestra que si se cambia de puerta, se gana en el 66,6% de los casos. El siguiente código lo demuestra, y además ilustra el uso de loops (función for) y funciones de generación de número aleatorios en R:
montehall<-function(repeticions,canvi)
{
cotxe<-trunc(runif(repeticions,1,3.99999999999999999999999999999999))
eleccio<-trunc(runif(repeticions,1,3.99999999999999999999999999999999))
res<-0
for(i in 1:repeticions)
{
if(cotxe[i]==eleccio[i])
{
if(cotxe[i]==1)
{
obrir<-trunc(runif(1,2,3.99999999999999999999999999999999))
}
if(cotxe[i]==2)
{
obrir<-trunc(runif(1,1,2.99999999999999999999999999999999))
if(obrir==2)
{
obrir=3
}
}
if(cotxe[i]==3)
{
obrir<-trunc(runif(1,1,2.99999999999999999999999999999999))
}
}
else
{
trampa<-seq(1:3)
obrir<-subset(trampa,trampa!=cotxe[i] & trampa!=eleccio[i])
}
if(canvi==T)
{
trampa2<-seq(1:3)
eleccio2<-subset(trampa2,trampa2!=obrir & trampa2!=eleccio[i])
}
else
{
eleccio2<-eleccio[i]
}
if(cotxe[i]==eleccio2)
{
res<-res+1
}
}
return(round(100*res/repeticions,digits=2))
}
Para realizar el experimento 1000 veces, cambiando siempre de puerta:
montehall (1000,T)
El resultado debería aproximarse a 66,6.
Documentación de funciones
Cada función debe ir acompañada de un archivo de ayuda que se presentará cuando el usuario teclee:
?nombrefunción
La forma más sencilla de hacerlo es crear un nuevo fichero llamado nombrefunción.Rd (por ejemplo interval.Rd) y escribir directamente la ayuda. Se aconseja que los ficheros de ayuda contengan siempre los mismos apartados (nombre de la función, parámetros, resultados, etc.), como se puede ver en el código fuente del paquete Traba.
Construcción de paquetes
Una vez tenemos el código fuente tanto de las funciones como de las ayudas que deseamos incluir en el paquete, debemos efectuar una serie de procedimientos para empaquetarlo de forma que sea directamente instalable en R. Dicho proceso se explica detalladamente en este pdf.
Exportación e integración con LaTeX
R permite la exportación de objetos para su inclusión en los documentos generados en LaTeX.
Sistemas pedestres
Una forma rápida de exportar objetos a LaTeX es mediante la librería xtable (tipo 3). Se trata de construir en R la tabla que se quiera exportar, y luego:
library(xtable) xtable(nombretabla)
Esto imprimirá por pantalla el código de la tabla en formato LaTeX, que podrá ser copiado y pegado a nuestro fichero .tex. Este sistema, aunque pedestre, evita tener que reconstruir las tablas cada vez que se cambian los datos, como ocurre en el (terriblemente pedestre) marco tradicional ($P$$ + M$ Excel + M$ Word).
La función presenta diversos posibles argumentos, consultables mediante:
library(xtable) ?xtable
Otra forma pedestre de exportar objetos a LaTeX es mediante la librería Hmisc (tipo 3). Su función latex genera archivos .tex que contienen el código LaTeX del objeto exportado:
library(Hmisc) latex(objeto,file="nombrearchivo.tex")
Si no se especifica una ruta, el archivo se generará en el working directory, que se puede conocer con:
getwd()
y cambiar mediante:
setwd("nuevaruta")
Sistema fino
Una forma mucho más adecuada y reproducible para integrar R con LaTeX es el sistema Sweave, que se explica con detalle en el artículo correspondiente.
Exportación a formato HTML
Sistemas pedestres
La librería xtable (tipo 3) es capaz de exportar objetos tabulares a formato HTML. Para ello debemos realizar los siguientes pasos:
library(xtable) nombre<-xtable(nombreobjeto) print(nombre,type="html")
Esto imprimirá por pantalla el código de la tabla en formato HTML, que podrá ser copiado y pegado a nuestro fichero .htm.
La función presenta diversos posibles argumentos, consultables mediante:
library(xtable) ?xtable
Otra forma pedestre de exportar objetos a formato HTML es mediante la librería Hmisc (tipo 3). Su función html genera archivos .html que contienen el código del objeto exportado:
library(Hmisc) html(objeto,file="nombrearchivo.html")
Si no se especifica una ruta, el archivo se generará en el working directory, que se puede conocer con:
getwd()
y cambiar mediante:
setwd("nuevaruta")
Sistema un poco más refinado (no mucho)
La librería R2HTML (tipo 3) contiene distintas funciones para la publicación de resultados en formato web. Su sintaxis, un poco compleja, se especifica a continuación a través de un ejemplo:
library(R2HTML)
HTMLStart(file.path("/home/acrida/Desktop"),HTMLframe=FALSE, Title="Probando",autobrowse=FALSE)
as.title("A ver cómo sale")
x <- 1
y<- 2
x+y
HTMLStop()
El ejemplo anterior va a generar el fichero index.htm en el escritorio de un sistema GNU/Linux.
Las múltiples posibilidades de estas funciones se pueden consultar en:
library(R2HTML) ?HTMLStart ?HTML
Sistema aún más refinado
La función Sweave, presentada en un apartado anterior por su integración con LaTeX, permite también la integración entre R y HTML de una forma totalmente limpia y reproducible. Es el sistema recomendado para la publicación de resultados en formato HTML. Explicado en el artículo correspondiente.
Sistema tan refinado que ya no sé ni como llamarle
I've seen things you people wouldn't believe (Roy Batty, Blade Runner).
Se trata de distintos sistemas que permiten que usuarios remotos interactuen por intranet o internet con nuestro sistema R (instalado en un servidor) a través de una interface web, es decir sólo usando su navegador. Esto permite a los usuarios remotos:
- Introducir datos en un formulario que hayamos diseñado nosotros, que pasará estos datos como argumentos a funciones de R que devolverán el output por pantalla al usuario remoto. Una de las formas más sencillas de realizarlo es mediante CGIwithR. Un ejemplo propio y otro un poco más avanzado.
- Analizar nuestras bases de datos introduciendo ellos mismos código de R a través del navegador. Se puede realizar, entre otros, mediante el paquete RPad, que permite además su uso como GUI si se realiza una instalación sólo local .
La instalación y puesto en funcionamiento de estos sistemas supera los objetivos de este artículo. Nuestra guía de configuración de un servidor contiene algunas pistas sobre la instalación de CGIwithR.
Las páginas web correspondientes a cada proyecto contienen abundante información sobre el tema.
Y aún más: integración con MediaWiki
En 2006 aparece una extensión de MediaWiki (el software que soporta la Wikipedia y esta WikiTraba entre muchos otros proyectos) que permite insertar sintaxis de R en las páginas de un proyecto wiki. Esta extensión está instalada en la WikiTraba y se explica en su Ayuda.
Un ejemplo casi-real
Se parte de dos tablas distintas en formato .dbf exportadas desde M$ Excel, y que se pueden descargar desde los siguientes enlaces:
- La tabla baixes.dbf (borrado por error, si alguien lo tiene que lo suba) contiene un registro para cada una de las bajas laborales concedidas durante 2004 en una empresa X (a leer en catalán). Puede haber más de una baja por individuo. La base contiene la identificación del trabajador, el tipo de baja (incapacidad transitoria o baja maternal) y las fechas de inicio y finalización de la baja.
- La tabla plantilla.dbf contiene los datos referentes a todos los trabajadores de la empresa X, con un sólo registro por individuo. Contiene la identificación del trabajador (permite el cruce con bajas.dbf), su fecha de nacimiento, el sexo y sus datos laborales (tipo de contrato, lugar de trabajo y categoría laboral).
Las tablas provienen de un ejemplo real, pero se han eliminados todos los datos que pudieran permitir identificar a los individuos.
El objetivo es conocer los factores asociados a las bajas laborales, tanto a nivel individual como de centro de trabajo. Para ello deberemos en primer lugar reestructurar los datos para conseguir tener dos tablas:
- la base individual donde deberán figurar todos los trabajadores con su edad, sexo, datos laborales, número de bajas durante 2004 y días totales de baja durante 2004.
- la base agregada donde deberán figurar todos los ambulatorios con su número de trabajadores, la edad media de los trabajadores, el número total de bajas solicitadas en 2004 y el total de días de baja que representaron.
Aunque hay muchos caminos para alcanzar este objetivo, utilizaremos en ocasiones el camino más largo para ir viendo distintas funciones de R. En la página de discusión de este artículo se intentará responder cualquier duda sobre el código. Se aconseja irlo ejecutando por partes para ir viendo como se comportan las funciones. Hay que remarcar que hay mucho código redundante que sólo pretende mostrar los cambios que se van introduciendo.
Se ruega notificar en la página de discusión cualquier error que se detecte.
1-Captura y modificación de baixes.dbf
- Capturamos, hacemos distintas comprobaciones y etiquetamos las variables y los valores
library(foreign)
baixes<-read.dbf("C:/rutacompleta/baixes.dbf")
names(baixes)
class(baixes$ID)
class(baixes$TIPUS)
levels(baixes$TIPUS)
baixes$TIPUS<-factor(baixes$TIPUS,labels=c("Incapacitat transitòria","Maternal"))
class(baixes$INICI)
class(baixes$FINAL)
library(Hmisc)
label(baixes$ID)<-"Identificació"
label(baixes$TIPUS)<-"Tipus de baixa"
label(baixes$INICI)<-"Data d'inici"
label(baixes$FINAL)<-"Data de finalització"
- Seleccionamos sólo las incapacidades transitorias, eliminando las bajas maternales
length(baixes$ID) baixes<-subset(baixes,baixes$TIPUS=="Incapacitat transitòria") length(baixes$ID) names(baixes) baixes$TIPUS<-NULL names(baixes)
- Seleccionamos sólo las bajas que empiezan antes del 1-1-2005 y borramos el resto
length(subset(baixes,is.na(baixes$INICI))$INICI)
length(baixes$INICI)
summary(baixes$INICI)
baixes<-subset(baixes,baixes$INICI<as.Date("2005-1-1"))
length(baixes$INICI)
summary(baixes$INICI)
- Dando un poco de vuelta para divertirnos, recodificamos los inicios anteriores a 1-1-2004 a esa fecha
for(i in 1:length(baixes$INICI)){
if(baixes$INICI[i]<as.Date("2004-1-1")){
baixes$INICI[i]=as.Date("2004-1-1")}}
length(baixes$INICI)
summary(baixes$INICI)
- Dando otro paseo, recodificamos las finalizaciones missing y las posteriores a 31-12-2004 a esa fecha
length(subset(baixes,is.na(baixes$FINAL))$FINAL)
for(i in 1:length(baixes$FINAL)){
if(is.na(baixes$FINAL[i])){
baixes$FINAL[i]=as.Date("2004-12-31")}}
length(subset(baixes,is.na(baixes$FINAL))$FINAL)
length(baixes$FINAL)
summary(baixes$FINAL)
for(i in 1:length(baixes$FINAL)){
if(baixes$FINAL[i]>as.Date("2004-12-31")){
baixes$FINAL[i]=as.Date("2004-12-31")}}
length(baixes$FINAL)
summary(baixes$FINAL)
- Calculamos la duración de las bajas
baixes$DURADA=baixes$FINAL-baixes$INICI+1 length(subset(baixes,is.na(baixes$DURADA))$DURADA) summary(baixes$DURADA) baixes$DURADA baixes$DURADA<-as.numeric(baixes$DURADA) baixes$DURADA
- Dando vuelta, buscamos bajas duplicadas y las borramos
baixes$DUPLICAT<-rep(1,length(baixes$ID))
table(baixes$DUPLICAT)
for(i in 2:length(baixes$ID)){
if(baixes$ID[i]==baixes$ID[i-1] & baixes$INICI[i]==baixes$INICI[i-1] & baixes$FINAL[i]==baixes$FINAL[i-1]){
baixes$DUPLICAT[i]=baixes$DUPLICAT[i-1]+1}}
table(baixes$DUPLICAT)
baixes<-subset(baixes,baixes$DUPLICAT==1)
table(baixes$DUPLICAT)
names(baixes)
baixes$DUPLICAT<-NULL
baixes$INICI<-NULL
baixes$FINAL<-NULL
names(baixes)
- Y ahora lo mismo (buscar y borrar duplicados) sin dar tanta vuelta
duplicat<-duplicated(cbind(baixes$ID,baixes$INICI,baixes$FINAL)) table(duplicat) baixes<-subset(baixes,duplicat==F) table(duplicated(cbind(baixes$ID,baixes$INICI,baixes$FINAL))) names(baixes) duplicat<-NULL baixes$INICI<-NULL baixes$FINAL<-NULL names(baixes)
- Ahora viene lo importante: reestructurar la base de datos para pasar a tener un solo registro por persona
length(baixes$ID)
table(duplicated(baixes$ID))
numero<-aggregate(baixes,by=list(baixes$ID),FUN=length)
length(numero[,1])
names(numero)
numero
numero$ID<-NULL
names(numero)<-c("ID","numero")
names(numero)
dies<-aggregate(baixes,by=list(baixes$ID),FUN=sum)
length(dies[,1])
names(dies)
dies
dies$ID<-NULL
names(dies)<-c("ID","dies")
names(dies)
- Y montamos la base definitiva de bajas
baixes2<-merge(numero,dies) class(baixes2) label(baixes2$ID)<-"Identificació" label(baixes2$numero)<-"Número de baixes durant 2004" label(baixes2$dies)<-"Total dies de baixa durant 2004" subset(baixes,ID==10) subset(baixes2,ID==10) baixes<-NULL baixes<-baixes2 baixes2<-NULL
2-Captura y modificación de plantilla.dbf
- Capturamos, hacemos distintas comprobaciones y etiquetamos las variables y los valores
library(foreign)
plantilla<-read.dbf("C:/rutacompleta/plantilla.dbf")
names(plantilla)
class(plantilla$ID)
class(plantilla$SEXE)
levels(plantilla$SEXE)
plantilla$SEXE<-factor(plantilla$SEXE,labels=c("Dona","Home"))
class(plantilla$NAIX)
class(plantilla$CATEG)
levels(plantilla$CATEG)
class(plantilla$CONTR)
levels(plantilla$CONTR)
class(plantilla$UP)
levels(plantilla$UP)
library(Hmisc)
label(plantilla$ID)<-"Identificació"
label(plantilla$SEXE)<-"Sexe"
label(plantilla$NAIX)<-"Data de naixement"
label(plantilla$CATEG)<-"Categoria laboral"
label(plantilla$CONTR)<-"Tipus de contracte"
label(plantilla$UP)<-"Unitat productiva"
- Nos quedamos sólo con los trabajadores de ambulatorio, identificables porque su código de puesto de trabajo empieza por las letras BR
length(plantilla$ID) plantilla<-subset(plantilla,substr(as.character(plantilla$UP),1,2)=="BR") length(plantilla$ID)
- Calculamos la edad
names(plantilla)
length(subset(plantilla,is.na(plantilla$NAIX))$NAIX)
plantilla$EDAT<-(as.Date("2004-7-1")-plantilla$NAIX)/365.25
plantilla$EDAT
plantilla$EDAT<-as.numeric(plantilla$EDAT)
plantilla$EDAT
length(subset(plantilla,is.na(plantilla$EDAT))$EDAT)
plantilla$NAIX<-NULL
names(plantilla)
- Buscamos duplicados sin encontrarlos
duplicat<-duplicated(plantilla$ID) table(duplicat) duplicat<-NULL
- Recodificamos la edad de forma diferente a como hemos hecho en baixes.dbf
library(car)
?recode
detach(package:Hmisc)
?recode
plantilla$EDATC<-recode(plantilla$EDAT,"55:hi=3;45:55=2;35:45=1;lo:35=0",T)
plantilla$EDATC
class(plantilla$EDATC)
levels(plantilla$EDATC)
plantilla$EDATC<-factor(plantilla$EDATC,labels=c("Menor de 35","35 a 45","45 a 55","Major de 55"))
plantilla$EDATC
library(Hmisc)
label(plantilla$EDATC)<-"Edat categoritzada"
3-Unimos las dos bases, datos individuales
- Vamos directamente al grano pero haciendo algunas comprobaciones prescindibles (prova)
length(plantilla$ID) length(baixes$ID) individual<-merge(plantilla,baixes,all.x=T) length(individual$ID) prova<-merge(plantilla,baixes,all.x=T,all.y=T) length(prova$ID) length(prova$ID)-length(individual$ID) length(subset(plantilla,is.na(plantilla$SEXE))$SEXE) length(subset(prova,is.na(prova$SEXE))$SEXE) prova<-NULL
- Ponemos ceros donde hay missings
length(subset(individual,is.na(individual$numero))$numero) detach(package:Hmisc) individual$numero<-recode(individual$numero,"NA=0") length(subset(individual,is.na(individual$numero))$numero) length(subset(individual,is.na(individual$dies))$dies) individual$dies<-recode(individual$dies,"NA=0") length(subset(individual,is.na(individual$dies))$dies)
4-Algunos análisis sin demasiado sentido en la base individual a la espera que alguien añada más
sexe<-prop.table(table(individual$SEXE))
sexe
barplot(sexe,col="orange",ylim=c(0,1))
prop.table(table(individual$CONTR,individual$SEXE),2)
tapply(individual$EDAT,individual$SEXE,mean)
boxplot(individual$EDAT~individual$SEXE,col=c("orange","blue"))
summary(glm(individual$numero~individual$EDAT+individual$SEXE+individual$CONTR,family=poisson))
5-Creación de los datos agregados, con un código manifiestamente mejorable
baixestotals<-aggregate(cbind(individual$UP,individual$numero,individual$dies),by=list(individual$UP),FUN=sum)
subset(individual,UP=="BR017")
subset(baixestotals,Group.1=="BR017")
baixestotals<-baixestotals[,c(1,3,4)]
names(baixestotals)<-c("UP","Baixes","Dies")
edatm<-aggregate(cbind(individual$UP,individual$EDAT),by=list(individual$UP),FUN=mean)
subset(individual,UP=="BR017")
subset(edatm,Group.1=="BR017")
edatm<-edatm[,c(1,3)]
names(edatm)<-c("UP","EdatM")
treballadors<-aggregate(individual,by=list(individual$UP),FUN=length)
subset(individual,UP=="BR017")
subset(treballadors,Group.1=="BR017")
treballadors<-treballadors[,c(1,2)]
names(treballadors)<-c("UP","Treballadors")
agregadapre<-merge(baixestotals,edatm)
agregada<-merge(agregadapre,treballadors)
names(agregada)
agregada
6-Algún análisis sobre los datos agregados
summary(glm(agregada$Baixes~agregada$EdatM+offset(log(agregada$Treballadors)),family=poisson))
Sistema de ayuda
El lenguaje S, y por tanto el sistema R, tiene tantas funciones que es imposible recordarlas todas. Conocer cómo buscar ayuda es imprescindible para un buen uso del programa.
Ayuda disponible en el propio sistema
Desde R podemos consultar la ayuda de todas las funciones contenidas en los paquetes que tengamos instalados. Hay tres formas de hacerlo:
- Si conocemos el nombre de la función que queremos utilizar pero queremos consultar sus argumentos u otros detalles:
?nombrefunción
- Si no conocemos el nombre de la función que queremos utilizar pero sabemos el nombre de la técnica (por ejemplo Kruskall-Wallis):
help.search("búsquedaarealizar")
help.search("kruskall")
- Si queremos navegar por todas las funciones que están disponibles en nuestro sistema:
help.start()
Lo cual abrirá nuestro navegador y nos permitirá navegar por todos los paquetes y funciones de los que disponemos. Además desde la misma ventana podremos accedes a distintos manuales oficiales.
Ayuda externa
La comunidad R posee una potente lista de correo que permite la rápida solución de dudas y problemas que puedan surgir durante el uso del programa. Sin embargo, sólo de forma excepcional será necesario realizar una pregunta, pues seguro que alguien se la ha planteado antes que nosotros. Por tanto, una forma rápida de resolver una duda es buscando en Google la palabra clave r-help (identificativo de la mayoría de listas de correo de la comunidad) seguido de la duda que queremos resolver.
Por ejemplo, si no sabemos cómo realizar una regresión logística y aplicamos la búsqueda sugerida, encontramos más de 3.000 resultados.
Los usuarios de Firefox pueden configurarse un marcador Quick search con la palabra clave r y la descripción http://www.google.com/search?q=r-help%20%s lo que permitirá realizar búsquedas en r-help escribiendo simplemente en la barra de direcciones r duda.
Este sistema puede ser muy útil cuando desconocemos qué técnica estadística aplicar, pero si ya sabemos por ejemplo que queremos usar una regresión logística pero no sabemos qué función utilizar per realizarla, lo más eficiente es buscar en R Help Center, que contiene las funciones y sus correspondientes ayudas de todos los paquetes disponibles en el CRAN.
Otros manuales
Manuales oficiales (en ingés)
Una web con un resumen sobre R y links a manuales muy buenos como este.
La web de Victor Moreno con distintos manuales y clases en castellano.
Otra clase en castellano.
Las diapos del último congreso de R (Viena 2004).
Reference card
Alguien puede revisarselas y ordenarlas por utilidad y darles algún comentario?
[3] Quizás me quedaría con esta...